
膝骨关节炎的全基因组相关性研究:现在和未来
引言
膝骨关节炎是一种常见的复杂疾病,由遗传和环境因素共同作用引起。许多研究都提供了骨关节炎遗传影响的有力证据,包括与骨关节炎相关的领养研究、双胞胎研究和孟德尔疾病研究。骨关节炎的遗传力估计在骨关节炎位点之间是不同的。膝关节骨性关节炎、髋关节骨性关节炎和腰椎骨性关节炎的遗传率分别为39%、60%和74%[1]。此外,表型和遗传相关性都存在于骨关节炎中,并且在某种程度上每个骨关节炎都被认为有一个共同的遗传背景。通过关联研究的方法来证明遗传因素的细节,迄今为止已经鉴定出许多OA易感基因。此项关联研究用于鉴定许多复杂疾病的易感基因。此项关联研究有两种方法。一种是候选基因研究,分析已知的候选基因。另一种是全基因组关联研究(GWAS),利用单核苷酸多态性(SNPs)分析整个基因组。GWAS于2002年在世界上首次用于心肌梗死 [2]。关于KOA,中岛等人[3]于2010年首次报告,迄今已有一些GWAS报告(表1) [5-9]。

Full table
在这篇中,我们回顾KOA基因研究的现状和问题,并介绍我们对早期KOA (eKOA)定义的方法。
骨关节炎遗传学研究的现状和问题
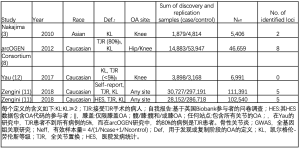
目前,遗传关联研究已经确定了约30个独立的OA易感性位点[10,11]。除GDF5外,所有基因位点均由GWASs鉴定;其中10个与KOA有关联。2010年,中岛等人首次报道了KOA GWAS,其中包括来自日本队列的899例病例和3396名人群对照(表2) [3]。其疾病定义是基于膝关节X线片[前后 (A-P) 视图]的凯尔格伦-劳伦斯(KL)等级。在使用980个病例和1418个对照进行重复实验后,他们鉴定了两个KOA易感基因。
Full table
2012年,一项名为arcOGEN的英国研究进行了一项GWAS,包括7410例骨关节炎病例(4144例膝骨关节炎病例)和11009例人群对照[8]。在对多达14883例病例和53947名对照进行重复实验和荟萃分析后,他们确定了8个具有全基因组意义的骨关节炎易感性位点,其中包括4个骨关节炎位点。此项英国研究的特点是,在研究的亚组中,OA的定义是不同的,在发现阶段使用的许多病例都是被认为患有OA的受试者,因为他们刚刚接受了全关节置换术(TJR)手术,而没有考虑影像学数据。
2017年,Yau等人对来自北美队列的3898例病例和3168名对照进行了KOA的GWAS[12]。他们使用KL分级和TJR来定义OA,但与arcOGEN研究不同的是,TJR患者在所有OA病例中的比例不到5%。尽管他们统一了队列中KL2级的解释来减少病例组的差异,但他们无法识别任何全基因组的显著基因位点。
2018年,Zengini等人使用英国生物库数据进行了一项OA GWAS[11]。在这项研究的发现阶段,他们使用了两种不同的OA定义:自我报告和医院事件统计(HES)。在对多达30727例病例和297191名对照进行重复实验和荟萃分析后,他们确定了10个全基因组的显著基因位点。这项研究的特点是,他们混合了不同部位(手、脊柱、臀部、膝盖等)的OAs,尽可能增加样本量。
GWAS强有力鉴定出的具有充分遗传证据的KOA易感基因只有9个(表1),这远远不能解释KOA的遗传性。此外,即使在同一种族的研究之间,也很少有基因在多个 GWAS 中达到全基因组显著的关联水平。可重复性低可能主要是因为这些OA GWAS的统计能力低。在GWAS中,有必要为多重测试的校正设定严格的标准[P值≤5×108 = 0.05/1,000,000(GWAS基因分型单核苷酸多态性的通常上限数)]。因此,要获得足够的GWAS统计能力,需要大量的样本。换句话说,迄今为止OA GWAS的样本量太小,无法检测到许多基因。在骨质疏松症和类风湿性关节炎等其他复杂疾病中,GWASs包括数万个病例,鉴定了100多个易感基因[13,14]。如此强大的GWAS也能识别许多KOA易感基因,尽管在实践中扩大样本量并不容易。
KOA GWAS的另一个问题是其模糊的表型定义。在许多流行病学和遗传学研究中,膝关节骨性关节炎通过膝关节平片的KL等级进行评估。虽然KL 2级或以上通常被定义为KOA,但KL 2级本身并没有明确的定义。科克霍夫等人[15]报告了在TREAT-OA联合会参与的28项研究中,人们使用了五种不同的KOA(KL 2级)定义。他们揭示了众多研究间KOA患病率差异的原因是定义的模糊性,并且由于KOA定义的统一,这些研究之间的患病率差异减小。因此,当我们使用KL等级来定义病例时,不同研究之间存在很多异质性。案例的这种异质性将降低GWAS分析的统计能力。
一个独特的表型提高了 OA GWAS 的能力
卡斯塔尼奥-贝当古等人[16]在2016年进行的髋关节骨性关节炎GWAS试验就是一个很好的例子,突出了表型定义的重要性。他们使用基于髋关节平片(A-P 视图)的最小关节间隙宽度 (mJSW) 作为软骨厚度的代表。然后,mJSW的GWAS在一个包含适度样本量(13013个)的发现集中进行了研究。在这个发现阶段,四个基因位点达到了全基因组显著性阈值。相比之下,在使用HES数据的英国生物库OA GWAS [11]中,没有基因位点在发现阶段达到全基因组显著性阈值,尽管其有效样本量(n=32280)相当大。HES是一个数据库,包含英格兰国家卫生服务医院所有入院和门诊预约的详细信息。因此,HES的数据是许多医生(专家)在日常实践中诊断的疾病名称的集合。专家通过KL分级[骨和(或)软骨特征的组合]以及日常实践中的临床主诉来诊断骨关节炎,这种模糊的标准导致了HES数据的异质性。另一方面,mJSW只关注软骨厚度,软骨是OA的主要受累组织,更加客观。此外,像mJSW这样的数量特征比二元特征拥有更多的信息。我们怀疑这种表型差异可能导致这两个GWAS之间的统计能力不同。
eKOA研究与骨关节炎遗传学研究的融合
近年来,人们提出了早期发现和治疗KOA的eKOA 概念,ESSKA(欧洲运动创伤学会膝关节手术和关节镜检查)于 2012 年发布了其定义[17]。在膝关节X线片被发现为关节间隙变窄之前,通过MRI或关节镜检查发现,软骨损伤是存在的。当伴随膝关节疼痛时,将其定义为eKOA。因此,这一定义更严格地评价了骨关节炎的主要受累组织——软骨的变化。这一定义的临床意义仍然未知,目前也没有与eKOA相关的遗传学研究。但是,我们认为检测软骨中的微小变化,即OA最早的变化,并且做出严格的定义是一个适用于遗传学研究中表型测定的概念。
在提出ESSKA的定义之前,我们已经开始了基于检测软骨早期变化的eKOA研究。侵入性和昂贵的检查方法,如核磁共振成像和关节镜检查,不适合需要大量样本的基因研究。于是我们采用了更简单和更便宜的美国评估方法。自2011年以来,我们正在根据这一分类进行队列研究。(详见Uchio和Kumahashi的章节)。我们根据美国的分类方法进行了流行病学研究,并阐明了美国定义下的普通人群 KOA 的分布和流行病学特征。 此外,我们展示了 KL 等级0级或1级的个体可以通过美国分类方法进行细分。我们希望基于我们美国评估的独特表型定义将改善 KOA GWAS。我们已经使用 RIKEN 中的队列数据启动了 GWAS项目。
为了揭示膝关节骨性关节炎的病因和发病机制,有必要利用骨科和基因组医学的知识对其进行探讨。随着 eKOA 证据的积累,建立更明确的 KOA 定义将提高 GWAS 的能力。越强大的GWAS将鉴定出越多的膝骨关节炎易感基因,这将有助于阐明膝骨关节炎的发病机制,建立有效的治疗和预防方法。
Acknowledgments
Funding: None.
Footnote
Provenance and Peer Review: This article was commissioned by the editorial office, Annals of Joint for the series “Early Osteoarthritis: Definition, Pathogenesis, Diagnosis, Management and Prevention”. The article has undergone external peer review.
Conflicts of Interest: All authors have completed the ICMJE uniform disclosure form (available at http://dx.doi.org/10.21037/aoj.2018.07.04). The series “Early Osteoarthritis: Definition, Pathogenesis, Diagnosis, Management and Prevention” was commissioned by the editorial office without any funding or sponsorship. YU served as the unpaid Guest Editor of the series. SI serves as an Editor-in-Chief of Annals of Joint from Mar 2016 to Feb 2021.The authors have no other conflicts of interest to declare.
Ethical Statement: The authors are accountable for all aspects of the work in ensuring that questions related to the accuracy or integrity of any part of the work are appropriately investigated and resolved.
Open Access Statement: This is an Open Access article distributed in accordance with the Creative Commons Attribution-NonCommercial-NoDerivs 4.0 International License (CC BY-NC-ND 4.0), which permits the non-commercial replication and distribution of the article with the strict proviso that no changes or edits are made and the original work is properly cited (including links to both the formal publication through the relevant DOI and the license). See: https://creativecommons.org/licenses/by-nc-nd/4.0/.
References
- Spector TD, MacGregor AJ. Risk factors for osteoarthritis: genetics. Osteoarthritis Cartilage 2004;12 Suppl A:S39-44.
- Ozaki K, Ohnishi Y, Iida A, et al. Functional SNPs in the lymphotoxin-alpha gene that are associated with susceptibility to myocardial infarction. Nat Genet 2002;32:650-4. [Crossref] [PubMed]
- Nakajima M, Takahashi A, Kou I, et al. New sequence variants in HLA class II/III region associated with susceptibility to knee osteoarthritis identified by genome-wide association study. PLoS One 2010;5:e9723 [Crossref] [PubMed]
- Miyamoto Y, Mabuchi A, Shi D, et al. A functional polymorphism in the 5' UTR of GDF5 is associated with susceptibility to osteoarthritis. Nat Genet 2007;39:529-33. [Crossref] [PubMed]
- Miyamoto Y, Shi D, Nakajima M, et al. Common variants in DVWA on chromosome 3p24.3 are associated with susceptibility to knee osteoarthritis. Nat Genet 2008;40:994-8. [Crossref] [PubMed]
- Evangelou E, Valdes AM, Kerkhof HJ, et al. Meta-analysis of genome-wide association studies confirms a susceptibility locus for knee osteoarthritis on chromosome 7q22. Ann Rheum Dis 2011;70:349-55. [Crossref] [PubMed]
- Day-Williams AG, Southam L, Panoutsopoulou K, et al. A variant in MCF2L is associated with osteoarthritis. Am J Hum Genet 2011;89:446-50. [Crossref] [PubMed]
- arcOGEN Consortium; arcOGEN Collaborators, Zeggini E, et al. Identification of new susceptibility loci for osteoarthritis (arcOGEN): a genome-wide association study. Lancet 2012;380:815-23.
- Valdes AM, Evangelou E, Kerkhof HJ, et al. The GDF5 rs143383 polymorphism is associated with osteoarthritis of the knee with genome-wide statistical significance. Ann Rheum Dis 2011;70:873-5. [Crossref] [PubMed]
- Cibrián Uhalte E, Wilkinson JM, Southam L, et al. understanding the genomic aetiology of osteoarthritis. Hum Mol Genet 2017;26:R193-201. [Crossref] [PubMed]
- Zengini E, Hatzikotoulas K, Tachmazidou I, et al. Genome-wide analyses using UK Biobank data provide insights into the genetic architecture of osteoarthritis. Nat Genet 2018;50:549-58. [Crossref] [PubMed]
- Yau MS, Yerges-Armstrong LM, Liu Y, et al. Genome-Wide Association Study of Radiographic Knee Osteoarthritis in North American Caucasians. Arthritis Rheumatol 2017;69:343-51. [Crossref] [PubMed]
- Karasik D, Rivadeneira F, Johnson ML. The genetics of bone mass and susceptibility to bone diseases. Nat Rev Rheumatol 2016;12:323-34. [Crossref] [PubMed]
- Okada Y, Wu D, Trynka G, et al. Genetics of rheumatoid arthritis contributes to biology and drug discovery. Nature 2014;506:376-81. [Crossref] [PubMed]
- Kerkhof HJ, Meulenbelt I, Akune T, et al. Recommendations for standardization and phenotype definitions in genetic studies of osteoarthritis: the TREAT-OA consortium. Osteoarthritis Cartilage 2011;19:254-64. [Crossref] [PubMed]
- Castaño-Betancourt MC, Evans DS, Ramos YF, et al. Novel Genetic Variants for Cartilage Thickness and Hip Osteoarthritis. PLoS Genet 2016;12:e1006260 [Crossref] [PubMed]
- Luyten FP, Denti M, Filardo G, et al. Definition and classification of early osteoarthritis of the knee. Knee Surg Sports Traumatol Arthrosc 2012;20:401-6. [Crossref] [PubMed]
(本译文仅供学术交流,实际内容请以英文原文为准。)
Cite this article as: Takuwa H, Uchio Y, Ikegawa S. Genome-wide association study of knee osteoarthritis: present and future. Ann Joint 2018;3:64.



